(english version below)
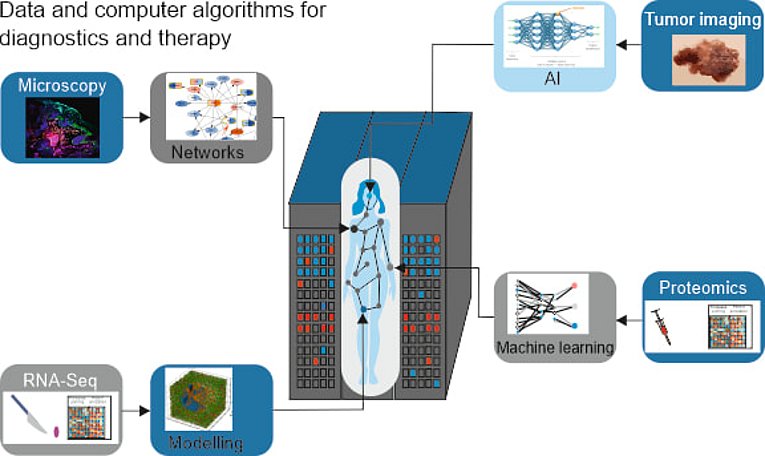
Unser internationales multidisziplinäres Team verfügt über mehr als ein Jahrzehnt Erfahrung in der computergestützten Modellforschung zur Aufklärung molekularer Mechanismen und zur Erkennung von Biomarker-Signaturen bei Melanomen und Krebsimmuntherapie (www.jveralab.net). Unsere Arbeitshypothese ist, dass multifaktorielle Krankheiten wie das Melanom nicht von einzelnen Genen, sondern von dichten und dysregulierten Netzwerken interagierender Gene, Proteine und RNAs gesteuert werden. Nur molekulare Hochdurchsatzdaten, fortgeschrittene Mathematik und Computer-Algorithmen können den Kern dieser Netzwerke aufklären und Targets für eine (personalisierte) Therapie vorschlagen.
Wir setzen auf Systemmedizin-basierende Ansätze, um den klinisch-tätigen Ärzten zu helfen, präzisere Diagnose- und Therapieentscheidungen zu treffen. Im Labor kombinieren wir Bioinformatik-Algorithmen, Computermodellierung und maschinelles Lernen, um klinische und molekulare Daten zu integrieren und Melanompatienten zu klassifizieren. Wir entwickeln Algorithmen der künstlichen Intelligenz für die Erkennung und Auswahl von Tumorepitopen und das Design von CAR-T-Zellen für die Krebstherapie (www.curatopes.com). Wir setzten pharmakogenomische Ansätze ein, um Medikamente umzuwidmen, die Patienten für eine Immuntherapie sensibilisieren und miRNA-basierte Krebstherapien zu entwickeln (www.synmirapy.net). Wir haben aus der Literatur abgeleitete Netzwerke von Genen und biomedizinischen Begriffen rechnerisch rekonstruiert, um molekulare Signaturen von Melanompatienten zu belegen und erklärbar zu machen.
Leitung
Prof. Dr. Julio Vera-González
Leiter "Laboratory of Systems Tumor Immunology"
Telefon: 09131 85-45876
Mitarbeitende
- Luca Musella
- Jimmy Retzlaff
Ausgewählte Drittmittelprojekte
Diagnostik mit Biomarkermustern aus extrazellulären Vesikeln des Plasmas mit Methoden der künstlichen Intelligenz (KI-VesD, gefördert durch das BMBF). Wir leisten einen Beitrag zur Klassifizierung von Patienten durch maschinelles Lernen auf der Basis klinischer und molekularer Daten. Webseite: https://kivesd-projekt.de
RNA-Verarbeitung für die Krebs-Immuntherapie (CANCERNA, EU HORIZON2020 Programm). Wir entwickeln computergestützte Algorithmen für die Erkennung und Auswahl von Tumorpeptiden zur Durchführung einer Melanom-RNA-Therapie. Webseite: https://cancerna.info
Systemmedizin des Melanoms und der Autoimmunität im Kontext der Immuntherapie (MelAutim, BMBF). Wir nutzten die Integration klinischer und molekularer Daten von Melanompatienten, um molekulare Signaturen zu erkennen, die dem Auftreten immunbedingter unerwünschter Wirkungen der Krebsimmuntherapie zugrunde liegen. Webseite: https://melautim.net
Identifizierung spezifischer Tumorantigene für die CAR-T-Zelltherapie gegen Aderhautmelanom durch RNA-Sequenzierung und computergestützte Modellierung (Hiege Stiftung). Wir nutzen RNA-Seq-Daten von Patienten und bioinformatische Algorithmen, um neue CAR-T-Zellen für therapieresistente Patienten mit kutanem und uvealem Melanom zu entwickeln
Ausgewählte wissenschaftliche Veröffentlichungen der Forschungsgruppe
ENQUIRE automatically reconstructs, expands, and drives enrichment analysis of gene and Mesh co-occurrence networks from context-specific biomedical literature (2025). Musella L, Afonso Castro A, Lai X, Widmann M, Vera J. PLoS Comput Biol. 21(2):e1012745. doi: 10.1371/journal.pcbi.1012745
Gene network-based and ensemble modeling-based selection of tumor-associated antigens with a predicted low risk of tissue damage for targeted immunotherapy (2024). Lischer C, Eberhardt M… Vera J. J Immunother Cancer. 12(5):e008104. doi: 10.1136/jitc-2023-008104
Artificial intelligence in cancer target identification and drug discovery (2022). You Y, Lai X, Pan Y, Zheng H, Vera J, Liu S, Deng S, Zhang L. Signal Transduct Target Ther. 7(1):156. doi: 10.1038/s41392-022-00994-0
A disease network-based deep learning approach for characterizing melanoma (2022). Lai X, Zhou J, Wessely A, Heppt M, Maier A, Berking C, Vera J, Zhang L. Int J Cancer. 150(6):1029-1044. doi: 10.1002/ijc.33860
Systems biology-based investigation of cooperating microRNAs as monotherapy or adjuvant therapy in cancer (2019). Lai X, Eberhardt M, Schmitz U, Vera J. Nucleic Acids Res. 47(15):7753-7766. doi: 10.1093/nar/gkz638
Laboratory of systems tumor immunology
Our international multidisciplinary team has over a decade of experience in computational model-driven research to elucidate molecular mechanisms and detect biomarker signatures in melanoma and cancer immunotherapy (www.jveralab.net). Our working hypothesis is that multifactorial diseases like melanoma cancer are not driven by single genes, but by dense and dysregulated networks of interacting genes, proteins and RNAs. Only high throughput molecular data, advanced mathematics and computational algorithms can elucidate the core of these networks and suggest targets for (personalized) therapy.
We implement systems medicine-based approaches to help clinicians make more precise diagnostics and therapy decisions. In the lab, we combine bioinformatics algorithms, computer modelling and machine learning to integrate clinical and molecular data and classify melanoma patients. We develop artificial intelligence algorithms for the detection and selection of tumor epitopes and the design of CAR-T cells for anti-cancer therapy (www.curatopes.com). We employed pharmacogenomics approaches to repurpose drugs that sensitize patients to immunotherapy, and to design miRNA-based anticancer therapies (www.synmirapy.net). We computationally reconstruct literature-derived networks of genes and biomedical terms to substantiate and make explainable molecular signatures from melanoma patients.
Selected third-party funded projects
Diagnostics with biomarker patterns from plasma extracellular vesicles using artificial intelligence methods (KI-VesD, funded by the BMBF). We contribute machine learning-based classification of patients based in clinical and molecular data. Webpage: https://kivesd-projekt.de/
RNA processing for anti-cancer immunotherapy (CANCERNA, EU HORIZON2020 program). We develop computational algorithms for the detection and selection of tumor peptides for implementing melanoma RNA therapy. Webpage: https://cancerna.info/
Systems medicine of melanoma and autoimmunity in the context of immunotherapy (MelAutim, BMBF). We exploited integration of clinical and molecular data from melanoma patients to detect molecular signatures underlying the onset of immune related adverse effects of cancer immunotherapy. Webpage: https://melautim.net
Identification of specific tumor antigens for CAR-T cell therapy against uveal melanoma by RNA sequencing and computational modelling (Hiege Stiftung). We exploit RNA-Seq data from patient and bioinformatics algorithms to design new CAR-T cells